UltraNuclease(M) ELISA Kit
产品说明书
FAQ
COA
已发表文献
UltraNuclease (M) ELISA Kit是一款针对Benzonase®核酸内切酶的ELISA残留检测试剂盒,该试剂盒能够特异性检测工艺中残留的Benzonase®核酸内切酶。
本试剂盒应用双抗夹心酶联免疫检测(ELISA)的实验原理,将Reference Standard(36702-B)和待测样品加入预包被抗Benzonase®核酸内切酶抗体的Pre-coated microtiter strips(36702-A),然后加入稀释后的生物素标记的酶检测抗体(36702-C),最后加入 Streptavidin-HRP(SA-HRP)(36702-D),形成抗体+抗原+抗体‐Biotin+ SA‐HRP 复合物,洗板后加入 TMB Substrate(36702–H)显色。TMB 在 HRP 酶的催化下由无色转化成蓝色并在Stop Solution(36702-I)的作用下最终转换成黄色。黄色的深浅与样品中被检测到的酶量呈正相关。
产品信息
|
货号 |
36702ES48 / 36702ES96 |
|
规格 |
48 T / 96 T |
|
检测范围 |
0.047-3 ng/mL |
|
检测方法 |
双抗夹心法 |
|
检测时长 |
3.5小时 |
|
灵敏度 |
0.044ng/mL |
|
回收率 |
75 – 125% |
|
板内差 |
<10% |
|
板间差 |
<15% |
实验数据
- 标曲数据
典型的参考标准曲线如下(以下标准曲线图仅供参考,应以同次实验标准品所绘标准曲线计算样本含量):
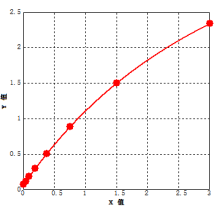
图1:典型标准曲线图
表1 典型标准曲线数据
|
标曲(ng/mL) |
OD |
平均OD |
|
|
3.000 |
2.353 |
2.328 |
2.340 |
|
1.500 |
1.502 |
1.500 |
1.501 |
|
0.750 |
0.891 |
0.886 |
0.888 |
|
0.375 |
0.492 |
0.513 |
0.503 |
|
0.188 |
0.291 |
0.309 |
0.300 |
|
0.094 |
0.181 |
0.191 |
0.186 |
|
0.047 |
0.120 |
0.118 |
0.119 |
|
0.000 |
0.081 |
0.073 |
0.077 |
- 检测流程图
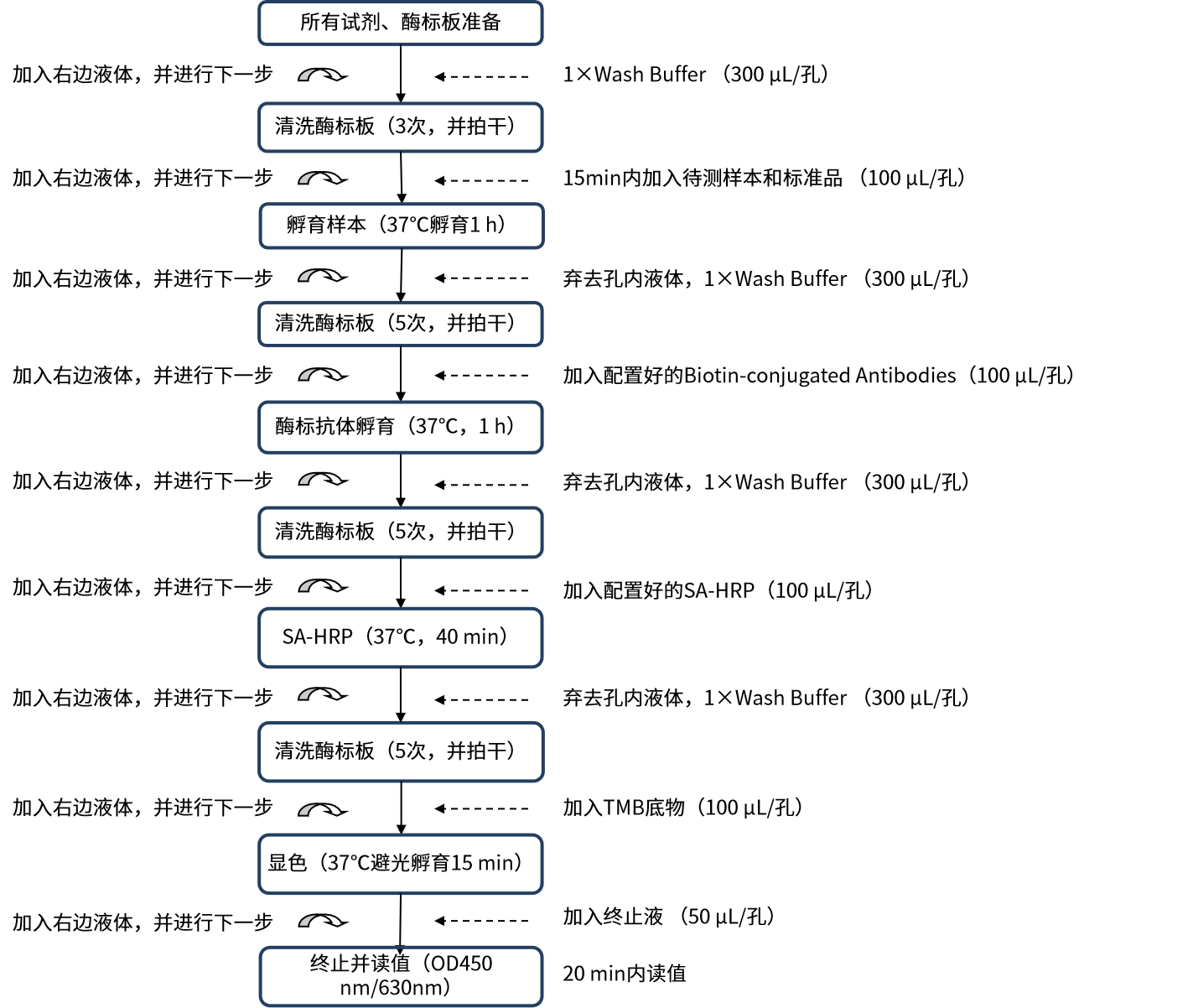
图2:实验步骤流程简图
Ver.CN20230605
UltraNuclease (M) ELISA Kit是一款针对Benzonase®核酸内切酶的ELISA残留检测试剂盒,该试剂盒能够特异性检测工艺中残留的Benzonase®核酸内切酶。
本试剂盒应用双抗夹心酶联免疫检测(ELISA)的实验原理,将Reference Standard(36702-B)和待测样品加入预包被抗Benzonase®核酸内切酶抗体的Pre-coated microtiter strips(36702-A),然后加入稀释后的生物素标记的酶检测抗体(36702-C),最后加入 Streptavidin-HRP(SA-HRP)(36702-D),形成抗体+抗原+抗体‐Biotin+ SA‐HRP 复合物,洗板后加入 TMB Substrate(36702–H)显色。TMB 在 HRP 酶的催化下由无色转化成蓝色并在Stop Solution(36702-I)的作用下最终转换成黄色。黄色的深浅与样品中被检测到的酶量呈正相关。
产品信息
|
货号 |
36702ES48 / 36702ES96 |
|
规格 |
48 T / 96 T |
|
检测范围 |
0.047-3 ng/mL |
|
检测方法 |
双抗夹心法 |
|
检测时长 |
3.5小时 |
|
灵敏度 |
0.044ng/mL |
|
回收率 |
75 – 125% |
|
板内差 |
<10% |
|
板间差 |
<15% |
实验数据
- 标曲数据
典型的参考标准曲线如下(以下标准曲线图仅供参考,应以同次实验标准品所绘标准曲线计算样本含量):
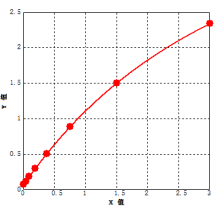
图1:典型标准曲线图
表1 典型标准曲线数据
|
标曲(ng/mL) |
OD |
平均OD |
|
|
3.000 |
2.353 |
2.328 |
2.340 |
|
1.500 |
1.502 |
1.500 |
1.501 |
|
0.750 |
0.891 |
0.886 |
0.888 |
|
0.375 |
0.492 |
0.513 |
0.503 |
|
0.188 |
0.291 |
0.309 |
0.300 |
|
0.094 |
0.181 |
0.191 |
0.186 |
|
0.047 |
0.120 |
0.118 |
0.119 |
|
0.000 |
0.081 |
0.073 |
0.077 |
- 检测流程图
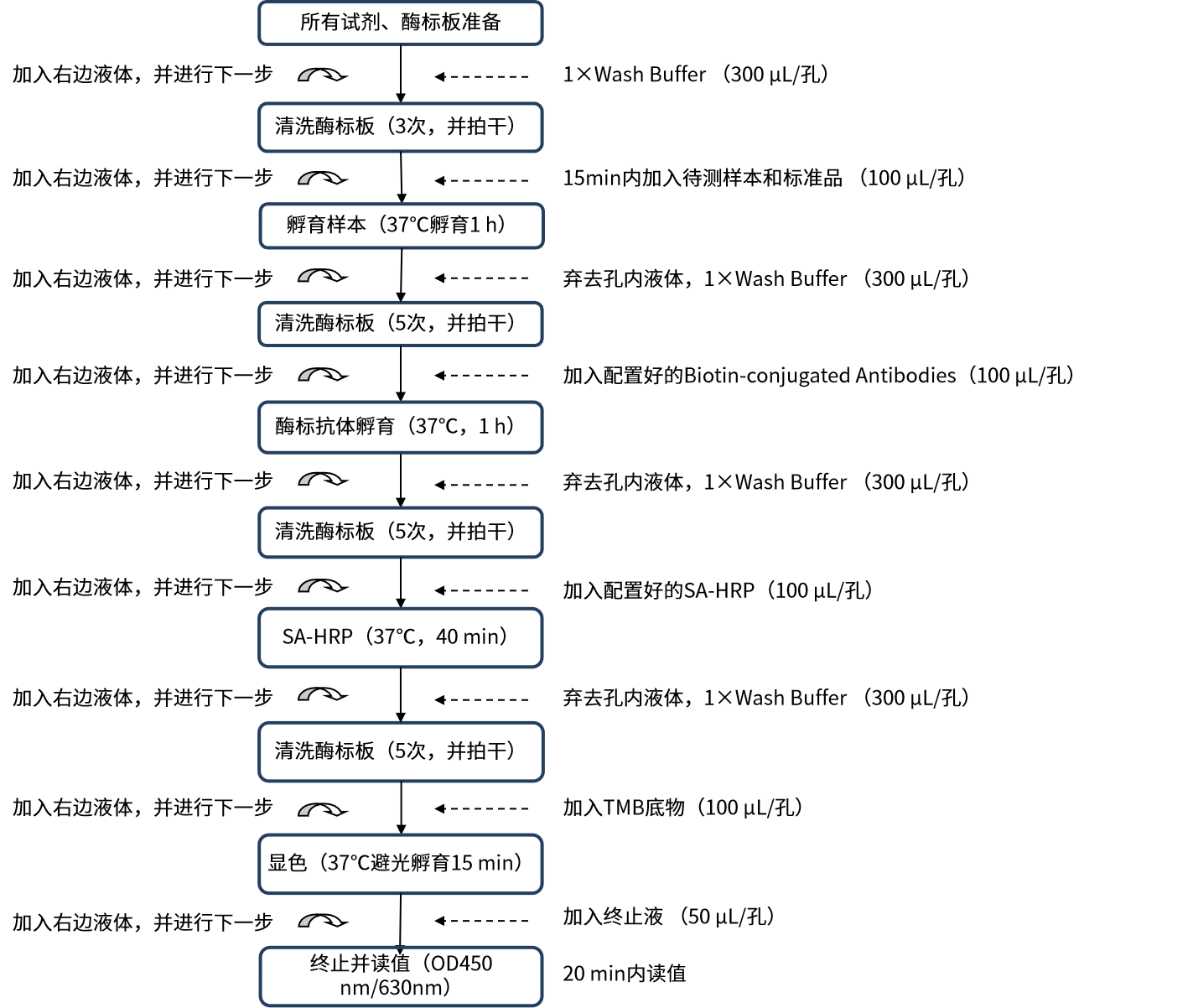
图2:实验步骤流程简图
Ver.CN20230605

暂无内容

暂无内容
